Dieses Interview ist zuerst im Agrarmagazin Landfreund erschienen und wurde über den landwirtschaftlichen Informationsdienst (LID) weiterverbreitet.
Herr Müller, Sie arbeiteten als Teil eines internationalen Forscherteams bei der erstmaligen vollständigen Sequenzierung des Tomaten-Genoms einer Wildtomate und der gezüchteten Tomatensorte "Heinz 1706" mit. Wie lange dauerte es um die 35 000 Tomaten-Gene zu entschlüsseln?
Lukas Müller: Das Tomatengenom-Projekt startete 2004, acht Jahre später publizierten wir die Resultate. Dabei wurden 850 Millionen Basenpaare analysiert. In den letzten zehn Jahren machten die Sequenzierungs-Technologien enorme Fortschritte. Wir mussten deshalb mehrmals praktisch von vorne beginnen, weil plötzlich viel schnellere Methoden zur Verfügung standen. Heute sprechen wir bereits von der dritten Generation von DNA-Sequenzierungsgeräten. Die alten Maschinen füllten noch das halbe Labor, der aktuell oft verwendete Nanopore-Sequenzer ist nur noch so gross wie ein USB-Stick und dazu viel leistungsfähiger.
Sie als Bioinformatiker speichern die Erbinformationen dann im Computer ab?
Wir erstellen in unserem Labor Datenbanken mit Genomen von verschiedenen Kulturen, die dann von Forschern und Zuchtfirmen genutzt werden können. Das Faszinierende dabei ist, dass sich die DNA von Pflanzen und anderen Lebewesen eigentlich sehr ähnlich wie ein Speicher im Computer verhält, beide Systeme funktionieren im Grunde digital.
"Es ist unethisch, wenn man solche Technologien und Informationen nicht nutzt"
Es gibt Leute, die in der Abbildung von DNA - dem Lebensplan der Geschöpfe - auf einem Computerbildschirm ein ethisches Problem sehen.
Die Frage für mich ist: Wie bringt man künftig genug Essen auf den Teller der zunehmenden Weltbevölkerung? Ich finde es unethisch, wenn man solche vorhandenen Technologien und Informationen nicht nutzt und die Leute verhungern lässt.
Was bringt es der Landwirtschaft, wenn man weiss, wie die DNA einer bestimmen Kultur aussieht?
In meiner Arbeit beschäftige ich mich zurzeit vor allem mit Maniok in Afrika. In diesem Projekt haben wir eine Datenbank aufgebaut mit über 30 000 verschiedenen Genotypen von Maniok. Dazu kommen 10 Millionen Phänotypen, mit verschiedenen äusseren Merkmalen, die auf dem Feld erfasst worden sind. Mit Software vergleichen wir die Daten der Genome und erhalten Kombinationen von Pflanzen, die sich für eine Kreuzung aufdrängen, um beispielsweise einen höheren Nährstoffgehalt zu erzielen.
"Bisher war die Zucht ein langsamer Prozess"
Die dabei angewendete genomische Selektion ist reine Züchtungstechnologie, die aber viel effizienter ist als herkömmliche Methoden. Bisher war die Zucht ein langsamer Prozess. Man musste die Pflanze wachsen lassen, sie betrachten und die äusseren Merkmale bestimmen. Dank den Sequenzierungs-Technologien wissen wir heute viel mehr über die Genome. Mit den immer leistungsfähigeren Computern können diese Millionen von Daten heute schnell verarbeitet und genutzt werden.
Wie lange dauert es heute, um beispielsweise ein Tomaten-Genom vollständig zu bestimmen?
Es dauert zwischen zwei Wochen und einem Monat. Früher brauchte es dafür Jahre und kostete Millionen, nun ist natürlich alles viel günstiger geworden.
Zur Person
Der Schweizer Lukas Müller lebt seit zwanzig Jahren in den USA und arbeitet als Bioinformatiker am Boyce Thompson Institute (BTI) der Cornell University in New York. Er studierte an der ETH Zürich Biochemie und doktorierte an der Uni Lausanne. Müller war Teil einer globalen Forschungsgruppe, die 2012 erstmals das vollständig entschlüsselte Genom einer Tomate publizierte. In seinem Labor sequenziert er Genome von landwirtschaftlichen Kulturen wie beispielsweise Tomaten, Kartoffeln oder Peperoni. Die erstellten Datenbanken stehen den Züchtern zur freien Nutzung zur Verfügung. Zurzeit beschäftigt sich Müller vor allem mit Projekten in Afrika zur Verbesserung von Sorten bei den Kulturen Maniok, Süsskartoffeln und Bananen.
Man kann nun also ohne viel Aufwand das Genom von jeder Sorte einer Kulturpflanze sequenzieren?
Theoretisch ja. Trotzdem lohnt es sich nicht, jede Sorte individuell zu sequenzieren, weil man ja eigentlich nur die Unterschiede zwischen den Sorten wissen möchte. Beim sogenannten Genotyping schaut man deshalb nur bestimmte Stellen im Genom an, wo man weiss, dass es viel Variabilität gibt.
Wie weiss man, welches Gen beispielsweise eine Resistenz gegen einen Pilz beinhaltet?
Im speziellen Fall von Resistenz-Genen in Pflanzen handelt es sich oft um Einzelgene, die man relativ leicht identifizieren und lokalisieren kann. Ein einzelnes Gen hat in diesem Fall einen grossen Effekt. Doch es braucht immer einen passenden genetischen Hintergrund, damit das Gen funktioniert. Manchmal funktioniert das identifizierte Gen gut, sehr oft aber eben auch nicht. Das ist alles nicht so einfach, wie es sich manchmal anhört. Noch interessanter wäre es zu wissen, wo das "Ertrags-Gen" liegt. Leider gibt es ein solches nicht, weil der Ertrag von hunderten von Genen gemeinsam bestimmt wird, was die Sache viel schwieriger macht.
"Leider gibt es kein Ertrags-Gen"
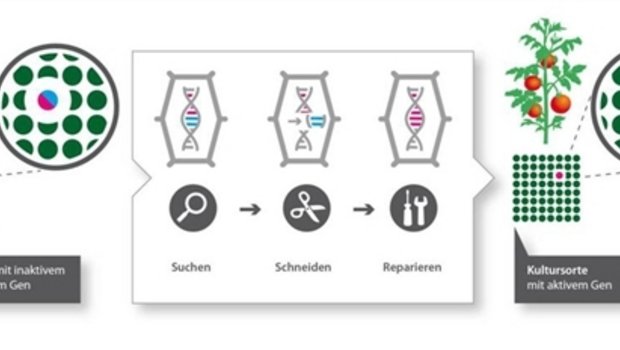
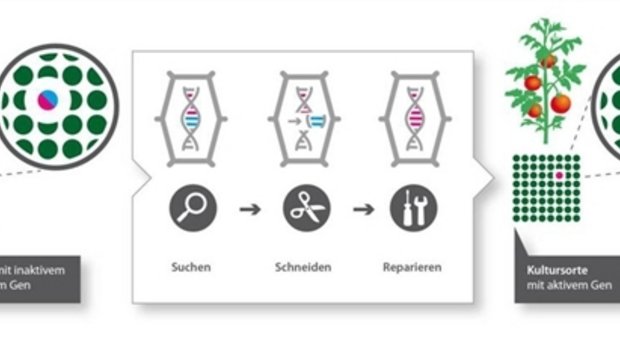
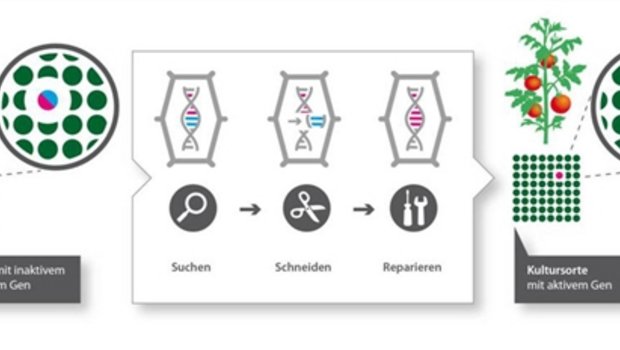
Trotzdem: Wenn das Resistenz-Gen bekannt ist, dann kann man es mit der Methode von CRISPR/Cas in anderen Organismen einsetzen?
Ja, aber man muss sehr vorsichtig sein, damit man nichts kaputt macht. CRISPR/Cas-Experimente wie bei den jüngst bekannt gewordenen chinesischen Zwillingen, mit Genom-Veränderungen in Embryonen, sind verantwortungslos. Die Technologie ist ja noch nicht vollkommen fehlerfrei und man muss immer noch sehr stark selektionieren. Wenn man das bei einer Pflanze macht und es geht etwas schief, ist das zudem weniger schlimm wie bei einem Menschen. Trotzdem braucht es für die Anwendung von CRISPR/Cas Regeln und Gesetze.
"Man muss sehr vorsichtig sein"
Der Europäische Gerichtshof hat CRISPR/Cas dem restriktiven Gentechnik-Gesetz unterstellt. Ist das für Sie nachvollziehbar?
Ich finde den Entscheid falsch. Die alten Methoden, bei denen Transgene eingepflanzt wurden, waren im Vergleich zu den heutigen neuen Technologien sehr ungenau und eigentliche Flickwerke, bei denen sogar Antibiotika-Resistenzen eingesetzt wurden. Aber bei CRISPR/Cas sieht man im Genom keine Unterschiede zu herkömmlichen Zuchtmethoden. Die Technologie ermöglicht sehr gezielte und spezifische Veränderungen. Sie ist zweifellos revolutionär.
Sie sehen keine Probleme bei CRISPR/Cas?
Aus wissenschaftlicher Sicht nicht. Ich sehe eher juristische Hindernisse, wie bereits bestehende Patentstreite, welche die Entwicklung hemmen und verteuern könnten. Das ist sicher eine problematische Entwicklung. Bisher war es üblich, dass Forschungsresultate der Öffentlichkeit zur Nutzung zur Verfügung gestellt werden. Unser Labor bekam die Problematik am eigenen Leib zu spüren, als wir plötzlich eine von unserer Uni entwickelte Sequenzierungstechnologie nicht mehr nutzen durften, weil sie in Holland patentiert worden war. Kommt dazu, dass Patente tendenziell die Abhängigkeit von Grossfirmen fördern.
"CRISPR/Cas könnte gegen Feuerbrand helfen"
Wie könnte der Schweizer Landwirt von CRISPR/Cas profitieren?
Konkret beispielweise bei der Bekämpfung von Feuerbrand beim Obst. Wir arbeiten zurzeit an einer ähnlichen Krankheit bei Zitrusfrüchten in den USA. Bei Ackerkulturen bin ich allerdings nicht auf dem aktuellsten Stand was CRISPR/Cas anbetrifft. Aber ich bin sicher, dass Zuchtfirmen weltweit an deren Weiterentwicklung arbeiten.
Erklärungen
DNA: Träger der Erbinformation
Genomsequenzierung: Bestimmung der Abfolge von Genen auf der DNA zur Entschlüsselung der Erbinformationen eines Organismus.
Genom: Gesamtheit der vererbbaren Informationen (Gene) eines Individuums (inklusive DNA).
Genotyp: Gesamtheit der Gene eines Organismus
Phänotyp: Äusseres Erscheinungsbild, Eigenschaften und Verhaltensmerkmale eines Organismus.
CRISPR/Cas: Biochemische Methode um DNA gezielt zu schneiden und zu verändern (Genschere).